細胞生化学実験 A
光学顕微鏡観察と画像処理技術
課題② 画像処理方法の演習
この実習課題では、下のサイトにある写真を使います。
コンピュータから接続して、画像は、各自のコンピュータにダウンロードして用います。
http://www.bio.chuo-u.ac.jp/nano/LM/Image/ImageJ_ex3.html
( http://www.bio.chuo-u.ac.jp/nano/ & http://www.bio.chuo-u.ac.jp/nano/
からもリンクされています)
|
☆この作業は、必ずしも実習室で行う必要はありませんが、コンピュータに画像解析ソフトImage Jがインストールされていることが必要になります。それには、以下の2つの方法があります。ただし、実習室以外で実施する場合でも、出欠を取るために、実習開始の時点で指定された教室に集合します。
・手持ちのコンピュータにImage Jをインストールし、用いる。 または、 ・手持ちのUSBメモリーにImage Jをインストールし、USB上から起動する。
☆ Image Jは、フリーソフトです。http://rsb.info.nih.gov/ij/ からOSを選んでダウンロードできます。インターネットにつながったコンピュータを使って以下の作業を実施します。
・ 上記アドレスより、Image J をダウンロードし、指示(英語表記)に従ってインストールします。インストール先は、各自の管理するフォルダー内の任意の場所です。大学の情報教育センターのコンピュータにはインストールできませんの、USBメモリー上で使用するようにします。 ・
インストール時に作成された ・ 下のようなツールバー(通常は画面右上)が表示されたら、実行可能な状態です。 |
*はじめに*
アップル社コンピュータ対応の画像処理ソフトとして開発されたNIH Imageは、その後、Windows版に対応したScion ImageやImage Jとして改良されて来ました。フリーのソフト(米国、NIH提供)で、画像計測や画像処理のための豊富な機能を持っているのが特徴です。ここでは、32-bitの画像データまで取り扱うことができ、生物分野でも汎用されているImage Jを使って、画像処理法の概要を演習します。<Q4~>は、質問や考察する事項ですが、レポートには、その答えを必ず記載します。
この演習では以下の画像データを使用します。(ア)~(ウ)の写真上でクリックして大きな画面を表示させた後に,右クリック(Webサイト上から)して、適宜、各自のコンピュータの任意フォルダーにダウンロードします。
(ア) Pleurosigma part(ファイル名:Pleurosigma_part.jpg)
(イ) SinePattern(ファイル名:SinePattern.bmp)
(ウ)Pleuropsigma(ファイル名:Pleurosigma_b.bmp)
(ⅰ) 基礎操作法の練習
a. 画像の読み込みと書き出し:上の(ア)の画像(ファイル名:Pleurosigma_part.jpg、ケイ藻細胞の光学顕微鏡写真、奥修博士提供)を開きます。このファイルをテキストデータとして書き出すには「File」→「Save as」→「Text image...」でファイル名を指定して出力できます。Jpgファイル(JPEG形式)の画像は、8ビット(256階調)で記録されているので、ここで出力したデータも、0~255の間の整数値(輝度データ)として出力されます。この機能を使って、画像⇒数値データの変換が自在にできることになります。逆の数値データ⇒画像の変換は「File」→「Import」→「Text image...」です。
b. 次に、ここで書き出したテキストデータのファイルをExcelで読み込みます。その中で、例えば16番目の列(P列) のデータを見ます。P列のデータをグラフ表示させると、画像の輝度データがどのように分布しているか、さらに、その変化の周期は、ピクセル数(画像の最小単位)で、およそ、どの程度になるか、わかります。このような方法で周期を調べることもできます。
c. 上のような数値をより正確に出すには、前回の実習「(ⅲ)-5」で修得した、「Straight line selection」を使って、観察したい方向を決め輝度データを読み込む方法、あるいは、単純にマウスで、位置を指定して、その座標を読み取って計算する方法など、いろいろな方法が考えられます。
d. 画像の明るさ・コントラストの調整:「Image」→「Adjust」→「Brightness/Contrast」で表示画像の明るさやコントラストを変えることができます。Brightnessとは、全体の明るさです。Contrastは、もっとも明るく表示する点と暗く表示する点との差に相当します。注意しなければならないのは、ここで変わるのは、表示方法だけという点です。もとの画像データ(256階調のデータ)にない数値情報は、どんなに輝度やコントラストを変化させても、あたらしく見えてくることはありません。最初に像を撮影したときの条件で、すべてが決まってしまいます。どのような顕微鏡写真を撮影したかで決まります。8-bitの輝度データのために、コントラストをあまり上げると、ざらざらした感触の画像となり、詳細が見えにくくなります。
e. 画像の回転、反転、擬似カラー表示などの加工:回転は「Image」→「Rotate」、反転は「Edit」→「Invert」、 擬似カラー表示は「Image」→「Lookup Tables」で選択し変えることができます。これも、表示方法の変更だけの意味しかありません。表現したい箇所を強調する時に使います。回転は、眼ではよくはわかりませんが、ピクセルの間の補間処理(ピクセルの間のデータを計算で予測する作業)を行いますので、実際は情報量として減少する方向にあります。
f. 画像のエッジを抽出する方法①:上の(ウ)の画像(ファイル名:Pleurosigma_b.bmp) を開き、「Process」→「Enhance Contrast 」の操作を行います。これによって、自動的にコントラストの増強加工を行います。もちろん、この操作でも、もとの画像の輝度データにないものが、見えてくる訳ではありません。
g. 画像のエッジを抽出する方法②:上と同じファイルを新しく開き、「Image」→「Duplicate」で同じ画像を別の名称で複製します。2つある画像の中で、片方を、「Process」→「Filters」→「Gaussian Blur (radius 20-30程度)」で処理します。この作業で、人工的にボケさせた画像を作成します。次に、その後、「Process」→「Image Calculator」で「Operation」を「Subtract」に選択した後に、上のボケ処理を行う前後2つの画像の間で減算処理(差の計算)を行うことができます。処理後、うまく表示できていなかったり、暗い像だったりしたら、上の(d)と同じ作業で、適切な明るさやコントラストを選択し、見やすくします。この減算処理の結果、どのようなことが起こるでしょうか<Q4>。なぜそのような変化が現れたかの考察も含めて、レポートに記載します。
h. このような画像処理で守らなければならない重要なルールがあります。顕微鏡の画像は、様々な処理を施す場合、最初のオリジナルの画像全体に同じ方法の画像処理を同等に実施することを心がけます。画像の一部、強調したい箇所だけを見やすくしたり、逆に、見せたくない箇所を削除したりするような操作は、技術的には容易に実施することができますが、サイエンスでは、真実をゆがめて伝えることに相当し、行ってはいけないねつ造行為となります。様々な画像処理を実施した場合、どのような処理を行ったか実験レポートには正確に記述します。
(ⅱ) 画像フーリエ変換
これまでの操作で、Image Jの中の簡単な操作方法を学習しました。実際の機能はさらに多種・多様で、研究・学習上で、より使いやすい機能を関連webサイトから見つけることができます。このセクションでは、2次元のフーリエ変換の演習を行います。上の(イ)の 画像(ファイル名:SinePattern.bmp)を開き、以下の操作を行います。これまで、すでに開いた他の画像があったら、それらはすべて閉じて構いません。
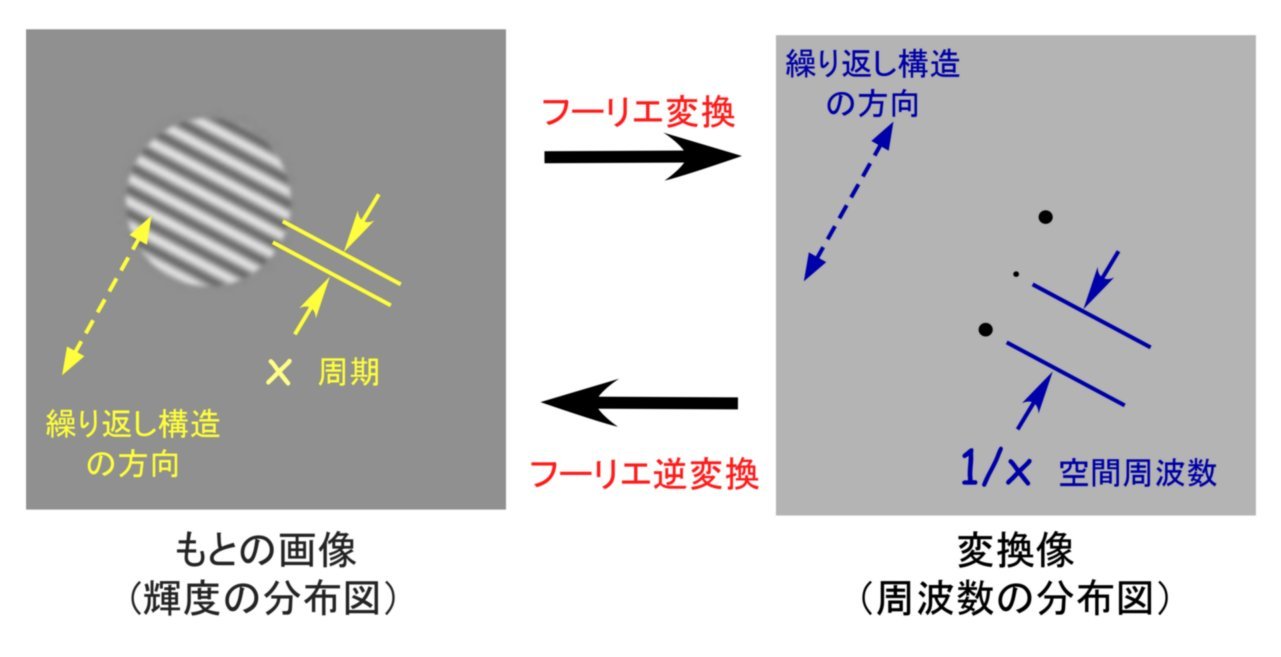
a. 2次元フーリエ変換:「Process」→「FFT」→「FFT」 と順番に実施します。ここで読み込んだ画像は、一定の周期で明暗が変化する縞模様です。上の操作では、別の新しい画像(FFT of SinePattern.bmpと名前が付いている画像)が作られるのがわかります。これは、SinePattern.bmpの画像の周波数を解析して、2次元マップとして表現したものです。このような画像をFFT画像(フーリエ変換後の画像、FFTはFast Fourier Transformの略です)と呼びます。このような操作を2次元画像のFFT変換、あるいは、FFT処理と呼びます。FFT変換とは何でしょうか?実際の作業の意味を理解する目的で、次の操作を行います。
b. Image Jのメニューの中で丸印のツールアイコン(Elliptical or brush selections)をまず選びます。次にFFT画像の中にある中で、1つの明るいスポット部分(中心以外にあるものの中から一つ選ぶ)を丸く囲み、「Edit」→「Cut」(又は,Ctrl+X)の操作で、この部分を削除します。その後、同じ箇所を「Edit」→「Invert」 (Cnrl+Shift+I)の 操作で黒く反転させます(プログラムの都合上必要な操作です)。この操作で,FFT画像の中にあるデータの一部が、完全に消去されたことになります。消去する場合、画面の中心を挟んでちょうど反対側にある対称な位置にあるスポットも、必ず同時に消去処理するようにします。この操作で、FFT画像の一部が欠落したものを人工的に作ることができます。
c. FFT画像の画面がactiveになっている(処理中の対象画像として選択されている)ことを確認し、Ctrl+A(全画面が選択される)を押した後に、「Process」→「FFT」→「Inverse FFT」を実施します。この操作は、上の(a)のまったく逆の演算を行います。別の画像(フーリエ逆変換像、Inverse FFT of SinePattern.bmpと名前が付いている画像)を表示する機能です。得られた新しい画像は、もとの画像から、上の(b)の操作で削除した情報が欠落したものに相当します。どの様に変化していますか<Q5>?(a)と(b)の作業を繰り返して、他のいろいろなスポットも削除する作業を試してみます。どのようなことが起こっているか、推測してレポートに記述します。
d. FFT画像の中の1つのスポットは、元の画像の上に周期的な模様(周期的な成分)があることを示しています。もとの画像の上では、その中心からそのスポットに向かう方向に、中心からの距離の逆数を周期とする繰り返しパターンがあることを意味しています。数学的には複雑な式で表現されますが、プリズムに光を通すとスペクトル(光の波長)に分けられるのと同じ様に、FFTは、信号の中の周期(周波数の逆数)の分布を調べるプログラムです。1次元ではなく、2次元なので、少し複雑にはなります。FFT画像の中心からの距離が周期の逆数ですが、これを「空間周波数」と呼びます。中心付近のスポットは、長い周期(低い空間周波数)のパターン、中心から離れた点は、短い周期(高い空間周波数)のパターンを反映しています。
e. 上の(ウ)の画像(ファイル名:Pleurosigma_b.bmp)を新しく開きます。同じようにFFT処理します。現れた代表的なスポット像(FFT画像)を使って、上の(b)→(c)の 操作を行います。その結果をもとに、このケイソウの細胞には、どのような周期パターン構造がありますか<Q6>?考察した結果をレポートに記述します。
f. 上の基礎演習(ⅰ)-gでは、2つの画像(ボケさせた画像とエッジを抽出した画像)を使いました。それぞれに、同様のFFT処理を行います。2つは、どのような点で違うでしょうか<Q7>?
g. 次の2つの画像、(エ)と(オ)は、ケイソウ Pleurosigmaを、異なる照明条件下で観察した結果を示しています。この場合、もとの細胞の構造は、ほぼ同じのはずですが、観察方法の違いによって、見える像が変わって来ます。どのような観察像の違いがあるでしょうか<Q8>?それぞれFFT画像変換の手法を作って比較した結果から、わかることをレポートに記述します。
(エ)暗視野照明条件下で観察したケイソウ(Webサイト上で右クリックするとダウンロードできます)
Dr. Roderich Zeiss ‘The Microscope and Its Revelations. 7. Auflage, London 1891’
(オ)光学顕微鏡写真(Webサイト上で右クリックするとダウンロードできます)

(ⅲ) 動画の解析
Image Jは、*.AVI形式で保存された動画の処理も豊富に備わっています。ここでは、動画から輝度データを読み取り、その時間的な変化を調べる例を演習します。
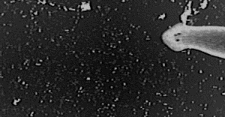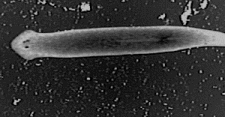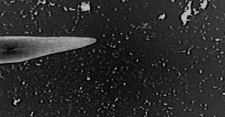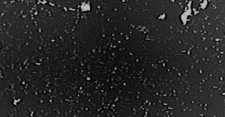
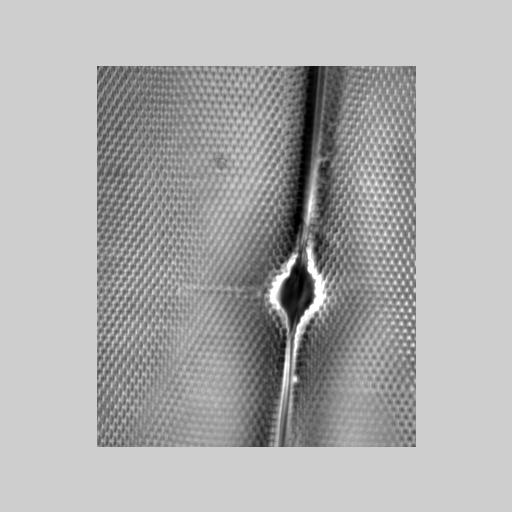
ここで用いるファイルは下の動画です。白い繊維状の構造があり、その上で運動する物体が観察されています。まず、ダウンロードして、各自のコンピュータに保存します。その後、Image Jで「File」→「Import」→「AVI…」で読み込んで使います。全部で200フレーム(動画の画像の枚数)のファイルです。コンピュータの性能上、実際にも、この程度の枚数(フレーム数)の動画が扱いやすい枚数です。非常に長いビデオ記録はコンピュータの負担を軽減するために、読み込み作業の時に、短い一部だけにして読み込みます。
a. 動画の表示:「Image」→「Stacks」→「Start Animation」によって動画の表示が始まります。動画は、2次元の映像(xy2次元データ)が、z方向(時間軸方向)へ順番に積み重なるようにして並んでいるものと見なすことができます。その積み重なったものをここでは「Stacks」と呼んでいます。動画は、xyzの3次元的な情報という考え方です。画像の下のバーを左右にスライド操作してもアニメーションの表示ができます。アニメーションの表示は画像の上でクリックすると停止します。
b. 時間軸方向への投影:「Image」→「Stacks」→「Z Project」によって、全時間の輝度データを重ねて表示します。xy面をz軸方向へすべて投影して重ねたものに相当します。この操作を少し応用して、画質の改善(加算平均)にも利用できます。下の手順を試します。
・ まず、読み込んだ動画(8bit-Stacks画像)を32bit-Stacksに変換します。「Image」→「Stacks」→「32-bit」で終了です。これは画像の内容は変化しませんが、256階調の輝度データを、32-bit = 4,294,967,295階調まで、大幅に表示枠を広げるための宣言です。ここで32-bitに変換できるものは、白黒の画像のみです。処理中に警告が出てきたら、はじめのAVI動画を読み込むときにレConvert to Grayscale(白黒画像)の設定で読み込むようにします。
・ 「Image」→「Stacks」→「Z Project」で、画像を重ねますが、このとき、Projection Typeで、Sum Slicesを選択します(Sum Slicesを選ぶと、場合によっては自動的に32-bit画像になります)。この作業で、全画像が加算されて新しい画像が作られます。Z軸方向(時間方向)への加算をしたことになります。①の(ⅲ)画像表示用ソフトの基本操作-dの操作、「Analyze」→「Histogram」、を使って、輝度データの分布を調べます。32-bitデータにする前後で、どのような違いがあるでしょうか<Q9>?
c. 輝度データの時間変化①:メニュー上のRectangular selections(![]() マーク)、あるいはPoint selections(
マーク)、あるいはPoint selections(![]() マーク)を使って、まず、解析したい領域、あるいは、点を選択します。その後、「Image」→「Stacks」→「Plot Z-axis Profile」と順番に実施すると、指定箇所の輝度データがどのように経時変化するかのグラフが表示されます。
マーク)を使って、まず、解析したい領域、あるいは、点を選択します。その後、「Image」→「Stacks」→「Plot Z-axis Profile」と順番に実施すると、指定箇所の輝度データがどのように経時変化するかのグラフが表示されます。
d. 輝度データの時間変化②:メニュー上のRectangular selections(![]() マーク)、あるいはStraight line selections(
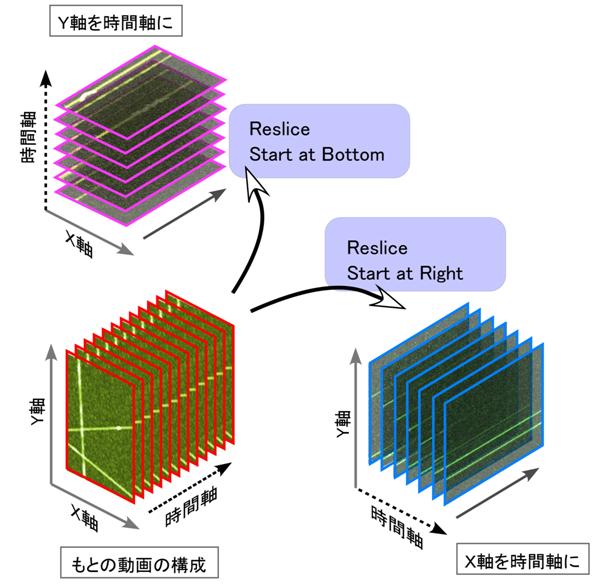
マーク)、あるいはStraight line selections(![]() マーク)を使って、まず、解析した領域、あるいは、線を選択します。その後、「Image」→「Stacks」→「Reslice」と順番に実施すると、指定箇所の輝度データがどのように変化するかの画像が表示されます。これは、動画をxyzの3次元画像と考えた場合に、そのxyのいずれかの軸を時間軸に置き換えて眺めたものと同じになります。なかなか難しそうな概念ですが、時間軸を変える作業は、下図を参考にして理解します。
マーク)を使って、まず、解析した領域、あるいは、線を選択します。その後、「Image」→「Stacks」→「Reslice」と順番に実施すると、指定箇所の輝度データがどのように変化するかの画像が表示されます。これは、動画をxyzの3次元画像と考えた場合に、そのxyのいずれかの軸を時間軸に置き換えて眺めたものと同じになります。なかなか難しそうな概念ですが、時間軸を変える作業は、下図を参考にして理解します。
e. 上の方法を参考にして、ここの例で見ている繊維状の構造の上で起こっている運動を解析します。どのような運動パターンが観察されるでしょうか<Q10>?
参考資料(Image Jその他の機能)
「Analyze」→「Measure」:面積、長さ、最大最小輝度などの表示
「Image」→「Adjust」→「Size」:画像の拡大・縮小
「Image」→「Cut, Copy, Invert ….」:画像のカット・コピー・反転・拡大・縮小など
「Process」→「Image Calculator」:画像データの間の四則演算
「Process」→「Math」:画像輝度データへの四則演算
「Process」→「Smooth, Enhance Contrast, Sharpen, Noise …」:画像の円滑化・コントラスト増強・エッジ強調など
「Plugins」→「Macro」:同じ様な繰り返し作業の記録、再生、作業の読み込みなど
参考サイト http://www.bio.chuo-u.ac.jp/nano/LM/Image/ImageJ_ex2.html
∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞
<実験が終わったら>
□ 用いたコンピュータの番号を実験ノートに記録します。
□ コンピュータの電源をOFF(電源を切る)にします。
□ コンピュータは、コンピュータ専用棚へ戻します。
□ ケーブル類、コネクタ類は、共通物品机に戻します。
□ 掃除当番(別の当番表参照)は、この実習で使った全机・周辺床の清掃をします。
∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞∞